EnGNet 1.0: Ensemble and Greedy Gene Networks
Descripción
Investigadores del Área de Lenguaje y Sistemas Informáticos de la Universidad Pablo de Olavide han desarrollado EnGNet 1.0, una aplicación informática que implementa una nueva metodología de explotación de datos de expresión genética denominada EnGNet, un nuevo algoritmo de dos pasos para la generación de redes de genes. Este programa es capaz de generar modelos biológicos empleando algoritmos de inteligencia artificial de alto rendimiento.
Necesidad o problema que resuelve
Las principales aplicaciones de esta nueva herramienta desarrollada en la Universidad Pablo de Olavide se centran en el campo de la Bioinformática y la Biomedicina. Así, por ejemplo con EnGNet 1.0 es posible llevar a cabo análisis biomédicos de diferentes procesos biológicos tales como análisis de biomarcadores, interacciones gen-gene entre otros. Actualmente la utilidad del software ha sido testada en el análisis del síndrome de estrés postraumático.
Y es que las redes genéticas se han convertido en una herramienta poderosa en el análisis integral de la expresión génica. Debido a la creciente cantidad de datos genéticos disponibles, los métodos computacionales para la generación de redes deben hacer frente a la llamada maldición de la dimensionalidad en la búsqueda de la fiabilidad de los resultados obtenidos. En este contexto, las técnicas de ensemble learning han mejorado significativamente la precisión de los resultados al combinar diferentes medidas, algoritmos o métodos. Por otro lado, las técnicas de optimización topológicas también son importantes en la reducción del tamaño de las redes, no sólo mejorando su arquitectura de red sino también manteniendo significancia biológica.
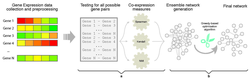
EnGNet 1.0 es una aplicación informática que implementa una nueva metodología de explotación de datos de expresión genética denominada EnGNet. EnGNet es un nuevo algoritmo de dos pasos para la generación de redes de genes. En primer lugar, el programa emplea una técnica de ensemble (“comité de máquinas”) para la generación de redes de co-expresión genética.
En segundo lugar, un algoritmo voraz optimiza tanto el tamaño como las características topológicas de la red. Este método no sólo es capaz de obtener redes fiables, sino que también mejora significativamente las características topológicas.
Aspectos Innovadores/Ventajas competitivas
EnGNet 1.0 es la primera implementación con interfaz visual de la metodología EnGNet para la generación de modelos biológicos. Como principales ventajas y aportaciones para la generación de redes genéticas por parte de la aplicación podemos destacar las siguientes:
- El método es capaz de superar las limitaciones de una única medida de co-expresión para generar la red genética, gracias a una estrategia de ensemble.
- El método también es capaz de realizar una optimización de la topología de la red final.
- Los resultados del método muestran su potencial en el campo del descubrimiento y caracterización de biomarcadores.
- La metodología seleccionada para la generación de los modelos (EnGNet) ha demostrado su superioridad respecto a los métodos estándares de generación de redes genéticas presentes en la literatura, por lo que supone un avance en el campo de estudio de la Bioinformática y en el estudio de procesos biológicos y enfermedades.
- EnGNet 1.0 ofrece una instalación stand-alone por lo que no se requiere el uso de internet o navegadores para poder ejecutar el mismo. Además, el diseño de la interfaz se ha realizado para buscar la mayor sencillez y facilidad de uso posible, por ello se ofrece, por ejemplo un panel de “Log” dentro de la misma aplicación donde el usuario podrá observar los diferentes mensajes que informen del estado de la ejecución y de una barra de progreso donde se podrá chequear el porcentaje realizado de la tarea.
Tipos de empresas interesadas
- Empresas del sector Bioinformático y Biomédico.
- Centros de investigación de los campos de la Bioinformática y la Biomedicina.
Nivel de desarrollo
Software inscrito en el Registro de Propiedad Intelectual. © Universidad Pablo de Olavide. Actualmente existe un prototipo testado en el análisis del síndrome de estrés postraumático.
Área tecnológica
Biotecnologia, Tecnologías de la información y de la Comunicación (Tic), Biomedicina y Salud Pública
Equipo de investigación
Intelligent Data Analysis (TIC239) > Más ofertas de este grupo
-
Autores: Francisco Antonio Gómez Vela, Fernando Miguel Delgado Chaves, Domingo Savio Rodríguez Baena, Miguel García Torres y Federico Divina.
-
Titular: © Universidad Pablo de Olavide.
Ficheros adjuntos
Contactar con la OTRI a través de nuestro formulario de contacto










